15. Modelos para la energía de gibbs de Exceso¶
Por lo regular 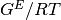 es una función de T, P y de la
composición, aunque para líqudios a presiones de bajas a moderadas es
una función muy débil de P. Por tanto, es usualmente despreciada la
dependencia de la presión de los coneficientes de actividad. En estos
términos, para los datos a T constante:
es una función de T, P y de la
composición, aunque para líqudios a presiones de bajas a moderadas es
una función muy débil de P. Por tanto, es usualmente despreciada la
dependencia de la presión de los coneficientes de actividad. En estos
términos, para los datos a T constante:
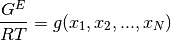
a T constante
La ecuación de Margules, es un ejemplo de dicha funcionalidad.
Un número de otras ecuaciones son de uso común para la correlación de
los coeficientes de actividad. En los sitemas binarios (especies 1 y 2),
la función representada con mayor frecuencia por una ecuación es
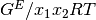 , la cual es factible expresar como una series de
potencias en
, la cual es factible expresar como una series de
potencias en 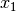
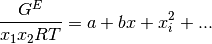
a T constante
Puesto que 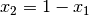 , la fracción mol
, la fracción mol 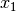 sirve
como la única variable independiente. una serie de potencias
equivalentes con ciertas ventajas se conoce como la expansión de
Redlisc/Kister
sirve
como la única variable independiente. una serie de potencias
equivalentes con ciertas ventajas se conoce como la expansión de
Redlisc/Kister
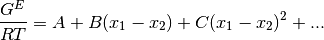
a T constante
en su aplicación son apropiados diversor truncamientso de esta serie y
en cada caso las expresiones especificas para 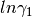 y
y
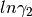 se genera con la ecuación
se genera con la ecuación
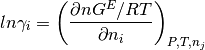
Cuando A=B=C=…=0, G^R/RT=0, ln _i=0 y la solución es ideal.
Si B = C = … = , entonces
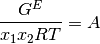
donde A es una constante para una termperatura dada. Las ecuaciones
correspondientes para 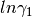 y
y 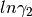 son:
son:
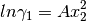
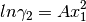
Es evidente la naturaleza simetríca de estas relaciones. Los
# -*- coding: utf-8 -*-
import numpy as np
def cal_NRTL(nC, T, Xi, Alfa, Aij):
#------------------------------------------------------------------------
s = (len(Xi),len(Xi))
Tao = G = np.zeros(s)
Tao = Aij / T
G = np.exp(-Alfa * Tao)
print ("\n", "Esta es la Matriz Tao = {0}".format(Tao), "\n")
print ("Esta es la Matriz G = {0}".format(G), "\n")
#------------------------------------------------------------------------
suma_1 = np.ones([nC, nC], dtype=np.float32)
suma_2 = np.ones([nC, nC], dtype=np.float32)
suma_11 = np.zeros([0, nC], dtype=np.float32)
suma_12 = np.zeros([0, nC], dtype=np.float32)
#------------------------------------------------------------------------
for j in range(nC):
for i in range(nC):
suma_1[i, j] = Tao[i, j] * G[i, j] * Xi[i]
suma_2[i, j] = G[i, j] * Xi[i]
suma_11 = suma_1.sum(axis=0)
suma_12 = suma_2.sum(axis=0)
#------------------------------------------------------------------------
print ("Esta es la Matriz suma1 = {0}".format(suma_1), "\n")
print ("Esta es la Matriz suma2 = {0}".format(suma_2), "\n")
print ("Esta es la Matriz suma11 = {0}".format(suma_11), "\n")
print ("Esta es la Matriz suma12 = {0}".format(suma_12), "\n")
#------------------------------------------------------------------------
A = suma_11 / suma_12
print ("miremos la matriz A = {0}".format(A), "\n")
#------------------------------------------------------------------------
num1 = np.zeros([nC, nC], dtype=np.float32)
den1 = np.zeros([nC, nC], dtype=np.float32)
num2 = np.zeros([nC, nC], dtype=np.float32)
den2 = np.zeros([nC, nC], dtype=np.float32)
for j in range(nC):
for i in range(nC):
num1[i, j] = G[j, i] * Xi[i]
den1[i, j] = G[i, j] * Xi[i]
num2[i, j] = Tao[i, j] * G[i, j] * Xi[i]
den2[i, j] = G[i, j] * Xi[i]
print ("Esta es la Matriz num1 = {0}".format(num1), "\n")
print ("Esta es la Matriz den1 = {0}".format(den1), "\n")
print ("Esta es la Matriz num2 = {0}".format(num2), "\n")
print ("Esta es la Matriz den2 = {0}".format(den2), "\n")
#------------------------------------------------------------------------
Z = np.zeros([nC, 1], dtype=np.float32)
W = np.zeros([nC, 1], dtype=np.float32)
lnGamma = np.zeros([nC, 1], dtype=np.float32)
ln = np.zeros([nC, nC], dtype=np.float32)
for i in range(nC):
Z[i, 0] = np.sum(den1[:, i])
W[i, 0] = np.sum(num2[:, i])
for j in range(nC):
for i in range(nC):
ln[i, j] = num1[i, j] / Z[i, 0] * (Tao[j, i] - W[i, 0] / Z[i, 0])
print ("Esta es la Matriz ln = {0}".format(ln))
#------------------------------------------------------------------------
for i in range(nC):
lnGamma[i, 0] = A[i] + sum(ln[:, i])
gamma_i = np.exp(lnGamma)
print ("Esta es la Matriz Z = {0}".format(Z), "\n")
print ("Esta es la Matriz W = {0}".format(W), "\n")
print ("Esta es la Matriz ln = {0}".format(ln), "\n")
print ("Esta es la Matriz lnGamma = {0}".format(lnGamma), "\n")
print ("Esta es la Matriz gamma_i = {0}".format(gamma_i), "\n")
#------------------------------------------------------------------------
return gamma_i
import numpy as np
#import NRTL_3
#------------------------------------------------------------------------
## Definiciones
#------------------------------------------------------------------------
# nC: Numero de componenetes de la mezcla
# T = Temperatura en K
# Xi = np.matrix([0.25, 0.25, 0.25, 0.25])
# Alfa =
# Aij =
#------------------------------------------------------------------------
# Alcohol Agua Acetato Acido
Alfa = np.array([[0.000, 0.2980, 0.3009, 0.1695],
[.2980, 0.0000, 0.2000, 0.2987],
[0.3009, 0.2000, 0.0000, 0.2000],
[0.1695, 0.2987, 0.2000, 0.0000]])
#------------------------------------------------------------------------
# Alcohol Acetato Agua Acido
Aij = np.array([[0.0000, 100.1, -144.8, 178.3],
[1447.5, 0.0000, 2221.5, 424.018],
[320.6521, 254.47, 0.0000, 214.55],
[-316.8, -110.57, -37.943, 0.000]])
#------------------------------------------------------------------------
nC = 4
T = 300.0
#------------------------------------------------------------------------
Xi_1 = float(eval(input("Fraccion molar 1: ")))
Xi_2 = float(eval(input("Fraccion molar 2: ")))
Xi_3 = float(eval(input("Fraccion molar 3: ")))
Xi_4 = float(eval(input("Fraccion molar 4: ")))
#------------------------------------------------------------------------
Xi = np.array([Xi_1, Xi_2, Xi_3, Xi_4])
sumar_Xi = sum(Xi)
Xi = Xi / sumar_Xi
#------------------------------------------------------------------------
print ("\n", "Composición Xi = {0}".format(Xi),"\n")
print ("Matriz Alfa = {0}".format(Alfa), "\n")
print ("Matriz Aij = {0}".format(Aij), "\n")
#------------------------------------------------------------------------
#CoeAct_1 = NRTL_3.NRTL(nC, T, Xi, Alfa, Aij)
coeficientes_actividad = cal_NRTL(nC, T, Xi, Alfa, Aij)
Fraccion molar 1: 0.2
Fraccion molar 2: 0.2
Fraccion molar 3: 0.3
Fraccion molar 4: 0.3
Composición Xi = [ 0.2 0.2 0.3 0.3]
Matriz Alfa = [[ 0. 0.298 0.3009 0.1695]
[ 0.298 0. 0.2 0.2987]
[ 0.3009 0.2 0. 0.2 ]
[ 0.1695 0.2987 0.2 0. ]]
Matriz Aij = [[ 0. 100.1 -144.8 178.3 ]
[ 1447.5 0. 2221.5 424.018 ]
[ 320.6521 254.47 0. 214.55 ]
[ -316.8 -110.57 -37.943 0. ]]
Esta es la Matriz Tao = [[ 0. 0.33366667 -0.48266667 0.59433333]
[ 4.825 0. 7.405 1.41339333]
[ 1.06884033 0.84823333 0. 0.71516667]
[-1.056 -0.36856667 -0.12647667 0. ]]
Esta es la Matriz G = [[ 1. 0.90535091 1.15631058 0.90416854]
[ 0.2374377 1. 0.22741016 0.65561563]
[ 0.72497794 0.84396296 1. 0.86672518]
[ 1.19601118 1.1163795 1.02561797 1. ]]
Esta es la Matriz suma1 = [[ 0. 0.06041708 -0.11162251 0.1074755 ]
[ 0.22912738 0. 0.33679447 0.18532856]
[ 0.2324657 0.21476325 0. 0.18595588]
[-0.37889633 -0.12343808 -0.03891502 0. ]]
Esta es la Matriz suma2 = [[ 0.2 0.18107018 0.23126212 0.18083371]
[ 0.04748754 0.2 0.04548203 0.13112313]
[ 0.21749339 0.25318888 0.30000001 0.26001754]
[ 0.35880336 0.33491385 0.30768541 0.30000001]]
Esta es la Matriz suma11 = [ 0.08269677 0.15174225 0.18625693 0.47875994]
Esta es la Matriz suma12 = [ 0.82378429 0.96917295 0.88442957 0.87197441]
miremos la matriz A = [ 0.10038643 0.15656881 0.21059555 0.54905277]
Esta es la Matriz num1 = [[ 0.2 0.04748754 0.14499559 0.23920223]
[ 0.18107018 0.2 0.16879259 0.2232759 ]
[ 0.34689316 0.06822305 0.30000001 0.30768541]
[ 0.27125058 0.19668469 0.26001754 0.30000001]]
Esta es la Matriz den1 = [[ 0.2 0.18107018 0.23126212 0.18083371]
[ 0.04748754 0.2 0.04548203 0.13112313]
[ 0.21749339 0.25318888 0.30000001 0.26001754]
[ 0.35880336 0.33491385 0.30768541 0.30000001]]
Esta es la Matriz num2 = [[ 0. 0.06041708 -0.11162251 0.1074755 ]
[ 0.22912738 0. 0.33679447 0.18532856]
[ 0.2324657 0.21476325 0. 0.18595588]
[-0.37889633 -0.12343808 -0.03891502 0. ]]
Esta es la Matriz den2 = [[ 0.2 0.18107018 0.23126212 0.18083371]
[ 0.04748754 0.2 0.04548203 0.13112313]
[ 0.21749339 0.25318888 0.30000001 0.26001754]
[ 0.35880336 0.33491385 0.30768541 0.30000001]]
Esta es la Matriz ln = [[-0.02437202 0.2723532 0.17045911 -0.33577991]
[ 0.03308712 -0.03230978 0.12046131 -0.12097954]
[-0.27191302 0.55496132 -0.07143436 -0.11726451]
[ 0.01408571 0.19496277 0.04953417 -0.18889984]]
Esta es la Matriz Z = [[ 0.82378429]
[ 0.96917295]
[ 0.88442957]
[ 0.87197441]]
Esta es la Matriz W = [[ 0.08269677]
[ 0.15174225]
[ 0.18625693]
[ 0.47875994]]
Esta es la Matriz ln = [[-0.02437202 0.2723532 0.17045911 -0.33577991]
[ 0.03308712 -0.03230978 0.12046131 -0.12097954]
[-0.27191302 0.55496132 -0.07143436 -0.11726451]
[ 0.01408571 0.19496277 0.04953417 -0.18889984]]
Esta es la Matriz lnGamma = [[-0.14872578]
[ 1.14653635]
[ 0.47961578]
[-0.21387103]]
Esta es la Matriz gamma_i = [[ 0.86180538]
[ 3.14727306]
[ 1.6154536 ]
[ 0.8074525 ]]
15.3 Modelos para la energía de gibbs de Exceso: UNIFAC¶
Ejemplo de implementación del modelo de actividad UNIFAC
U = 5;
m = 2; #Este es el número de moléculas en la mezcla
#g = 3; #Este es el número de grupos funcionales en la mezcla
g = 7;
#T = 331.15 # K
#T = 328
# Etanol - n-Hexano
#xj = [0.332 , 0.668]
#xj = [0.383 , 0.617]
###################################################
# Agua - Isoamil alcohol - ácido acético
# H2O CH3 CH2 CH OH COOH COOCH3
v1 = [1 0 0 0 0 0 0]'; # Agua
v2 = [0 2 2 1 0 0 1]'; # Isoamil acetato
v3 = [0 1 0 0 0 1 0]'; # Ácido acético
v = [v1' ; v2' ; v3']';
v = [v1' ; v3']';
###################################################
# Agua - Isoamil acetato - ácido acético
# H2O CH3 CH2 CH OH COOH COOCH3
R = [0.9200 0.9011 0.6744 0.4469 1.0000 1.3013 1.9031]';
Q = [1.4000 0.8480 0.5400 0.2280 1.2000 1.2240 1.7280]';
###################################################
# Agua - Isoamil alcohol - Ácido acético
# H2O CH3 CH2 CH OH COOH COOCH3
a = [0 300 300 300 -229.1 -14.09 72.8700;...
1318 0 0 0 986.5 663.5 232.100;...
1318 0 0 0 986.5 663.5 232.100;...
1318 0 0 0 986.5 663.5 232.100;...
353.5 156.4 156.4 156.4 0 199 101.100;...
-66.17 315.3 315.3 315.3 -151 0 -256.300;...
200.800 114.800 114.800 114.800 245.400 660.200 0 ];
###################################################
A = exp(-a./T);
####################################################
for j = 1 : 1 : m
r(:,j) = sum(R.*v(:,j));
end
r;
####################################################
for j = 1 : 1 : m
q(:,j) = sum(Q.*v(:,j));
end
q;
####################################################
for j = 1 : 1 : m
J(:,j) = r(1,j)*xj(1,j)/sum(r.*xj);
end
J;
####################################################
for j = 1 : 1 : m
L(1,j) = q(1,j)*xj(1,j)/sum(q.*xj);
end
L;
####################################################
li = 5.*(r - q) - (r - 1);
####################################################
lnYCi = log(J./xj) + 5.*q.*log(L./J) + li - (J./xj).*(sum(xj.*li));
#lnY1C = log(J(1,1)/xj(1,1)) + 5*q(1,1)*log(L(1,1)/J(1,1)) + li(1,1) - (J(1,1)/xj(1,1))*(xj(1,1)*li(1,1) + xj(1,2)*li(1,2))
#lnY2C = log(J(1,2)/xj(1,2)) + 5*q(1,2)*log(L(1,2)/J(1,2)) + li(1,2) - (J(1,2)/xj(1,2))*(xj(1,1)*li(1,1) + xj(1,2)*li(1,2))
###################################################
# Coeficiente de actividad residual del grupo (k)
# en la molecula (i) ######################
# Fracción molar del grupo funcional (k)
# en la molecula (i)
######################################################
######################################################
for i = 1 : 1 : m #Molécula (i)
for k = 1 : 1 : g #Grupo funcional (k)
xg(k,i) = v(k,i)./sum(v(:,i));
end
end
xg;
######################################################
for i = 1 : 1 : m #Molécula (i)
for k = 1 : 1 : g #Grupo funcional (k)
Lg(k,i) = Q(k,1)*xg(k,i)/sum(Q.*xg(:,i));
end
end
Lg;
#mor
######################################################
for i = 1 : 1 : m #Molécula (i)
for k = 1 : 1 : g #Grupo funcional (k)
ST(k,i) = sum(Lg(:,i).*A(:,k));
end
end
ST = ST';
######################################################
for i = 1 : 1 : m #Molécula (i)
for k = 1 : 1 : g #Grupo funcional (k)
if i == 1
STa(k,:) = (Lg(:,i)'.*A(k,:));
elseif i == 2
STa(k + g,:) = (Lg(:,i)'.*A(k,:));
elseif i == 3
STa(k + 2*g,:) = (Lg(:,i)'.*A(k,:));
end
end
end
STa;
######################################################
for i = 1 : 1 : m #Molécula (i)
for k = 1 : 1 : g #Grupo funcional (k)
if i == 1
lnTg(i,k) = Q(k,1).*(1 - log(ST(i,k)) - sum(STa(k,:)./ST(i,:)));
elseif i == 2
lnTg(i,k) = Q(k,1).*(1 - log(ST(i,k)) - sum(STa(k+g,:)./ST(i,:)));
elseif i == 3
lnTg(i,k) = Q(k,1).*(1 - log(ST(i,k)) - sum(STa(k+2*g,:)./ST(i,:)));
end
end
end
#lnT(1,k) = Q(k,1).*(1 - log(STg(1,k)) - sum(STga(k,:)./STg));
lnTg;
####################################################
#mor
######################################################
# Coeficiente de actividad residual del grupo (k)
# en la mezcla ######################
# Fracción molar del grupo funcional (k)
# en la mezcla
######################################################
for i = 1 : 1 : m #Molécula (i)
STq(:,i) = sum(v(:,i)*xj(:,i));
end
STq = sum(STq);
######################################################
for k = 1 : 1 : g #Grupo funcional (k)
xs(k,:) = (sum(v(k,:).*xj))./(STq);
end
xs;
######################################################
for k = 1 : 1 : g #Grupo funcional (k)
Lgs(k,1) = Q(k,1)*xs(k,1)/sum(Q.*xs);
end
Lgs;
######################################################
######################################################
for k = 1 : 1 : g #Grupo funcional (k)
STg(k,:) = sum(Lgs.*A(:,k));
end
STg = STg';
######################################################
for k = 1 : 1 : g #Grupo funcional (k)
STga(k,:) = (Lgs'.*A(k,:));
end
STga;
######################################################
for k = 1 : 1 : g #Grupo funcional (k)
lnT(1,k) = Q(k,1).*(1 - log(STg(1,k)) - sum(STga(k,:)./STg));
end
lnT;
####################################################
#Coeficiente de actividad Residual
for i = 1 : 1 : m #Molécula (i)
lnYRi(:,i) = sum(v(:,i).*(lnT' - lnTg(i,:)'));
lnYRi;
#Coeficiente de actividad total
lnYi = lnYCi + lnYRi
Yi = np.exp(lnYi)
- Bibliografía
| [1] | Michael L. Michelsen and Jorgen M. Mollerup. Thermodynamics Models: Fundamentals & Computacional aspects. Denmark. Second Edition. 2007. |
| [2] | Introductory Chemical engineering thermodynamics. J. Richard Elliott , Carl T. Lira. Prentice Hall (2012) |